Que materiais eu preciso para construir um modelo de DNA para um projeto de ciências?
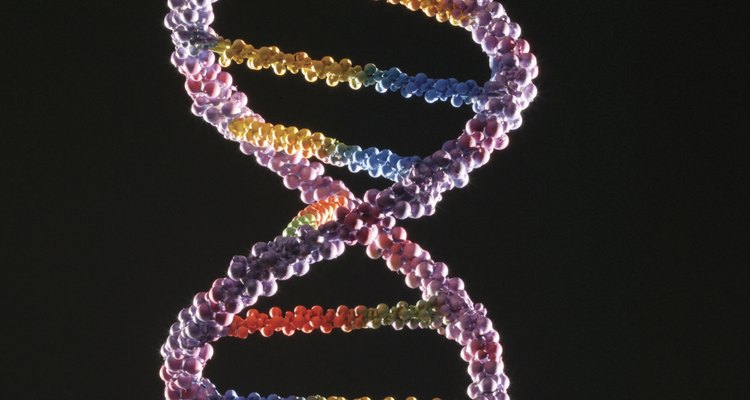
Comstock/Comstock/Getty Images
Construir uma réplica do modelo de dupla hélice do DNA é um projeto popular para ser feito em feiras de ciências. Construir o seu modelo de DNA é relativamente simples de se fazer e pode ser feito com itens baratos e fáceis de se encontrar. Impressione e informe os seus colegas de classe, professores ou avaliadores em uma feira de ciências com sua recriação de uma dupla hélice de DNA, enquanto lhes fornece informações sobre a estrutura do DNA.
Bolas de isopor
Um pacote com cerca de 100 bolas de isopor com 2,5 a 5 cm de diâmetro pode ser usado para representar as seis moléculas que se combinam para codificar uma informação genética no DNA - citosina, guanina, adenina, timina, pentose de açúcar e fosfato. Note que se você comprar um kit de construção de um modelo de DNA, ele irá conter o número necessário de bolas de isopor.
Tinta e pincéis
Oito cores diferentes são necessárias para diferenciar as moléculas em uma estrutura de DNA. 15 das suas bolas de isopor representam a pentose de açúcar, enquanto outras 15 representam as moléculas de fosfato - essas duas moléculas formam a armação, ou os degraus da escada da dupla hélice, e sempre ocorrerão na extremidade exterior do modelo. Você deve separar outras 60 bolas, dividi-las em grupos de 10 e pintá-las de cores diferentes para demonstrar claramente cada uma das seis moléculas remanescentes. Um pôster pode ser desenhado como legenda para explicar a molécula que cada cor representa, caso você esteja construindo seu modelo para uma apresentação ou feira de ciências.
Palitos de dente com dupla terminação
As linhas do seu modelo de DNA vão alternar entre quatro e duas moléculas. As linhas com quatro moléculas sempre terão uma pentose de açúcar em cada ponta, enquanto todas as linhas de duas moléculas são apenas duas moléculas de fosfato, sem nenhuma base de nucleotídeo presa a ele. Use palitos de dente de terminação dupla para juntar as bolas de isopor umas às outras. Os palitos de dente não representam parte alguma do DNA, mas são essenciais para segurar o modelo. Use os palitos de dente para criar a forma de dupla hélice do seu modelo, que deve começar quatro degraus abaixo da fila central e terminar quatro degraus acima do centro do modelo.
Suporte de madeira e cola
Construa o seu modelo de dupla hélice usando um suporte de madeira. O melhor suporte de madeira para esse propósito é uma base quadrada pesada que tenha um mastro se estendendo verticalmente do seu centro. Use isso para ancorar seu modelo de dupla hélice, prendendo os pareamentos de bases de nucleotídeos ou os palitos de dentre que ligam esses pareamentos ao mastro usando cola.
Referências
Recursos
Sobre o Autor
I have been involved in coaching and administration of youth soccer with the Herts FA for several years. I have many years experience with the technical side and equipment of soccer, cricket, rugby, snooker and poker. I studied the health and fitness and dietary side of competitive sport while at University. Currently, I am not ready for on-camera opportunities, but this could change with access to training and equipment.
Créditos Fotográficos
Comstock/Comstock/Getty Images